穆德添 万凌云 章瑶 韦树根 陆英 付金娥 田艺 潘丽梅 唐其
(1. 湖南农业大学园艺学院, 长沙 410128;
2. 广西壮族自治区药用植物园,南宁 510023)
钩藤是茜草科植物钩藤Uncaria rhynchophylla(Miq.)Miq.ex Havil、大叶钩藤Uncaria macrophyllaWall.、毛钩藤Uncaria hirsutaHavil.、华钩藤Uncaria sinensis(Oliv.)Havil.或无柄果钩藤Uncaria sessilifructusRoxb.的干燥带钩茎枝[1],为我国传统的大宗中药材。钩藤中含有生物碱、黄酮、三萜等活性成分[2],能够治疗阿尔兹海症、高血压、心率失调等症[3-5],有重要的药用价值。目前,钩藤是“钩藤片”“天麻钩藤颗粒”“脉君安”等诸多著名中成药的主要原料药和中药配方的常用药,钩藤的市场需求现已呈逐年递增的趋势。钩藤的研究目前集中于钩藤的药理药效[6]、有效成分的分离与鉴定[7-8]等方面,而对于钩藤中次生代谢产物的生物合成分子机制的研究甚少。阐明钩藤生物碱生物合成途径,梳理与解析途径中相关基因的分子调控机制,将为钩藤的开发与利用或分子育种奠定基础。
实时荧光定量PCR(real-time quantitative PCR,RT-qPCR)是目前基因表达水平检测的最常用方法。因其灵敏度高、特异性强、操作简单,在当前生物学研究中发挥着重要的作用[9]。然而,其结果的准确性和可靠性取决于RNA 的质量、反转录效率、引物的特异性以及管家基因的稳定性[10-11]。开展基因表达研究是发掘功能基因的重要手段之一,研究基因表达水平往往需要筛选出特定条件下最佳内参基因[12]。表达稳定的管家基因,往往被用于RT-qPCR分析的看家基因。植物RT-qPCR 中常用的管家基因包含18S 核糖RNA(18S)、3-磷酸甘油醛脱氢酶(GAPDH)、α 微管蛋白基因(TUA)、β 微管蛋白基因(TUB)、延伸因子基因(EF1-α)、肌动蛋白基因(Actin)、S-腺苷甲硫氨酸脱羧酶(SAM)等。在诸多研究中发现,不同植物间管家基因的表达稳定性也不同。如涂冬萍[13]等研究发现18S是在不同时期的有籽和无籽罗汉果果实中均稳定表达的内参基因;
杨婷[14]等研究发现ACT7和PP2A是铁十字海棠斑叶发育过程中最适的内参基因;
郭茜茜[15]通过实验发现GAPDH是钩藤蒴果中表达稳定性最高的基因。目前,还未见钩藤不同部位管家基因筛选的研究,因此选择合适的内参基因进行校准对目的基因表达模式分析具有重要的作用。
钩藤中的生物碱类型主要为萜类吲哚生物碱(terpenoid indole alkaloids, TIAs),其中最主要的为钩藤碱及异钩藤碱。钩藤生物碱在植物中含量较低,通过传统的育种来进行遗传改良周期长、成本高。阐明钩藤生物碱生物合成途径,获得控制其生物碱含量的关键酶基因,通过分子育种或者合成生物学手段实现钩藤有效成分的绿色生物制造是其新的途径。吲哚类生物碱的生物合成涉及多条代谢途径。来自非甲羟戊酸路径(“non-mevalonate” pathway,MEP/DOXP pathway)的异戊烯二磷酸(isopentenyl diphosphate, IPP)[16-17], 经 由 环 烯 醚 萜 途 径(iridoid pathway)的香叶醇-10 羟化酶(geraniol-10-hydroxylase, G10H)、马钱子酸甲基化酶(loganic acid methyltransferase, LAMT)和开环马钱子苷合成酶(secologanin synthase, SLS)等酶催化,生成开环马钱子苷(又称开联番木鳖苷)(secologanin)[18-21]。再和色胺途径的终产物色胺(tryptamine)经由异胡豆苷合成酶(strictosidine synthase, STR)催化作用形成异胡豆苷(strictosidine)[22],再进入TIAs 生物合成路径,这个过程中涉及到酶的编码基因称为参与TIAs 合成的“上游途径基因”。
本课题组完成了钩藤的基因组测序,发现了大量钩藤TIAs 生物合成途径的基因。基于基因组和转录组数据筛选出钩藤12 个候选管家基因,利用不同部位的钩藤材料鉴定这些候选管家基因的稳定性。进而利用筛选出的最适管家基因分析TIAs 合成相关的“上游途径基因”表达模式,为后续钩藤生物碱合成的关键酶基因功能验证和TIAs 生物合成途径的阐明奠定基础。
1.1 材料
于湖南农业大学国家中药材生产(湖南)技术中心剪取钩藤不同部位(根、茎钩、叶片、蒴果)植物材料,经广西药用植物园韦树根研究员鉴定为钩藤Uncaria rhynchophylla(Miq.)Miq.ex Havil。剪取后的材料用灭菌水洗净,吸干水分后,各组织迅速切片,液氮速冻,保存于-80℃冰箱备用。用于含量检测的钩藤样品,经过60℃烘干后粉碎,过60目筛,备用。
1.2 方法
1.2.1 基于LC-MS 技术的钩藤生物碱含量的测定 称取钩藤干燥粉末1 g,加入50 mL 质量浓度为80%的甲醇,称定锥形瓶总质量,置于50℃超声提取40 min,提取完毕后用提取液补充至超声前锥形瓶总质量,摇匀后取溶液过0.22 μm 有机滤膜,液相小瓶备用。采用Agilent 6530 LC-MS 系统(Agilent Technologies, Palo Alto, CA, USA)开展定量分析。分离条件使用反相色谱柱(SB C18柱, 2.1 mm × 100 mm, 1.8 μm, Agilent Technologies),0.1%甲 酸 水(A相)和0.1%甲酸乙腈(B 相)为溶剂体系,采用合适的梯度洗脱:0-20 min, 10%-70% B,流速为0.15 mL/min。色谱柱恒温35℃,进样量0.2 μL。测定得到不同浓度条件下钩藤生物碱标准物质的质谱响应峰面积,制备标准曲线,对不同部位中钩藤生物碱成分进行定量分析。
1.2.2 钩藤不同部位总RNA 的提取和cDNA 的合成 选取钩藤不同部位的植物材料,各取50-100 mg 样品浸润于液氮中,充分研磨后利用多糖多酚植物RNA 提取试剂盒进行总RNA 提取。采用微量分光光度计测定总RNA 的纯度和浓度。使用1%琼脂糖凝胶电泳仪检测总RNA 的完整性。用600 ng RNA 进行cDNA 第一条链的合成,操作步骤参照PrimerScriptTMRT reagent Kit with gDNA Eraser(Perfect Real Time)试剂盒(TaKaRa,日本)说明书,产物放置-20℃ 保存备用。
1.2.3 管家基因和钩藤生物碱合成相关基因的筛选及引物设计 基于钩藤基因组数据库筛选12 个候选管家基因分别为18S、GAPDH、Actin6、EF1-β、TUB、CYP、EF1-α、PAL、RNA L13、SAM、cdc73、TUA。另外,基于“基因组+全长转录组+代谢组”共表达分析方法筛选钩藤生物碱生物合成途径中的“上游途径基因”,分别为G8H、8-HGO、IS、CYP76A26、7-DLGT、7-DLH、LAMT、SLS、AS、AnPRT、IGPS、TSA、TSB、TDC、STR。使用Primer 5 软件设计荧光定量引物(表1,表2)并由生工生物工程(上海)股份有限公司合成。

表1 钩藤生物碱上游合成途径中关键酶基因RT-qPCR 的引物序列Table 1 Primer sequences for key genes in U. rhynchophylla alkaloids upstream synthesis pathway for RT-qPCR

表2 钩藤12 个候选管家基因RT-qPCR 的引物序列和扩增系数Table 2 Primer sequence and amplification parameters for 12 candidate reference genes of U. rhynchophylla in RT-qPCR
1.2.4 候选管家基因引物特异性测定和RT-qPCR 反应 将不同部位样品的 cDNA 模板原液等量混合,依次稀释5 倍,设置5 个浓度梯度,分别为模板原液的50、5-1、5-2、5-3、5-4倍。通过RT-qPCR 反应,获得每个管家基因在不同梯度下的Ct 值,生成标准曲线,计算斜率(k)、线性相关系数(R2)、以及利用公式E=(10-1/k-1)×100%计算管家基因的扩增效率。
使 用qTower3G 实 时 荧 光 定 量PCR 仪(analytikjena 公司,德国)进行 qPCR 反应,20 μL反应体系:2X SYBR GreenPro TaqHS premix 10 μL(Vazyme,中国),cDNA 2 μL,上下游引物各0.4 μL,RNase free water 7.2 μL。cDNA 模板是原液稀释6 倍,扩增反应程序为:95℃,30 s;
95℃,5 s;
60℃,30 s;
共40 个循环,每个样品进行3 次重复检测,反应结束后由软件生成熔解曲线。根据熔解曲线判断引物的扩增特异性,并且对荧光定量PCR 所得到的数据进行整理,获得12 个候选管家基因的Ct 值,并用2-△△Ct分析定量结果。
1.2.5 管家基因稳定性评价 使用GeNorm[23]、Normfinder[24]和BestKeeper[25]3 个软件及△Ct 程序[26]和RefFinder 在线网站[27]分析候选管家基因在钩藤不同部位的表达稳定性。GeNorm 和Normfinder 软件使用前,需将Ct 值转化为Q 值[28]再进行后续分析。转化公式:Q=2Ctmin-Ctsample(Ctsample为候选管家基因在不同实验组中的Ct 值,Ctmin为候选管家基因在所有实验组中最小Ct 值)。BestKeeper 和△Ct 可对候选管家基因的Ct 值直接计算分析。根据以上软件程序的分析结果结合RefFinder 在线网站(http://blooge.cn/RefFinder/)对12 个候选管家基因的稳定性进行综合评估,筛选出在钩藤不同部位中最优的管家基因。根据筛选出的管家基因,对钩藤生物碱合成上游途径(吲哚途径和环烯醚萜苷类途径)中的关键酶基因在钩藤不同部位进行表达分析,每个样品进行3 次技术重复并利用IBM SPSS Statistic 22 软件进行差异显著性分析。
2.1 钩藤生物碱含量分析
钩藤碱和异钩藤碱在钩藤蒴果和茎钩中含量较高,在叶片含量较低,几乎不在根中积累(图1)。

图1 钩藤不同部位中钩藤碱(A)和异钩藤碱(B)的含量Fig. 1 Rhynchophylline and isorhynchophylline contents from U. rhynchophylla different issues
2.2 样品总RNA质量检测引物特异性分析
使用1%的琼脂糖电泳对提取的总RNA 进行完整性检测。结果表明,钩藤4 个部位的总RNA 提取完整性较好,28S、18S 和5S 条带清晰明亮(图2)。进一步使用超微量分光光度计检测RNA 浓度和纯度,结果显示,4 个部位(根、茎钩、叶片、蒴果)的 总RNA 浓 度 分 别 为159.0 ng/μL、111.1 ng/μL、469.1 ng/μL、110.0 ng/μL,所 有 样 品 的OD260/280在1.90-2.10 范围内,OD260/230在1.80-1.90 区间。以上结果表明,样品总RNA 的完整性较好,纯度高,可用于后续实验。
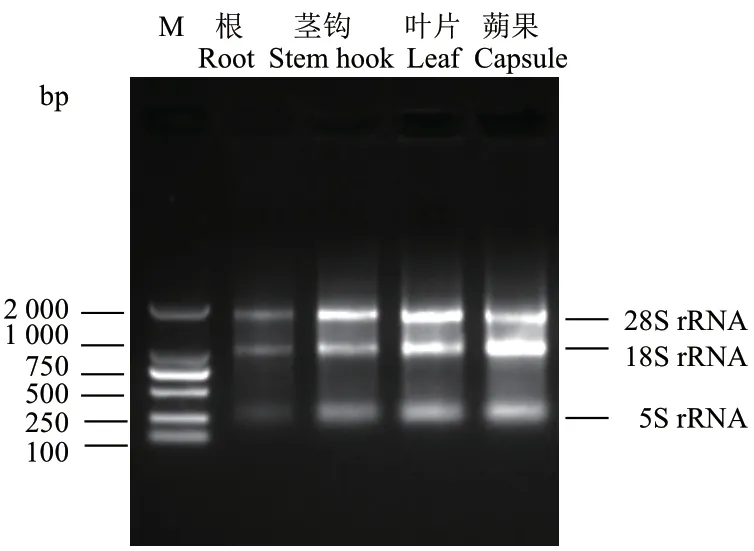
图2 钩藤不同部位总RNA 琼脂糖凝胶电泳Fig. 2 Agarose gel electrophoresis of total RNA from different tissues of U. rhynchophylla
2.3 候选管家基因的RT-qPCR引物特异性分析
以5 个浓度梯度的cDNA 为模板,对12 个候选管家基因的引物进行RT-qPCR 反应,利用下机数据绘制每个管家基因的标准曲线。结果表明,12 个管家基因的相关系数R2均在0.99 以上,各引物的扩增效率介于0.87%-1.09%之间,说明cDNA 模板量与对应的Ct 值呈现较好的线性关系,且12 个候选管家基因的熔解曲线均为单一熔解峰,曲线平滑,不存在引物二聚体(图3),表明引物特异性强,同一样品重复性好,可进行后续实验。

图3 12 个候选管家基因的RT-qPCR 引物的熔解曲线Fig. 3 RT-qPCR melting curves of 12 candidate house-keeping genes
2.4 候选管家基因表达丰度分析
Ct 值可直接反应基因的表达丰度,Ct 值越小则表明该基因的表达丰度越高。由图4 可知,在钩藤4 个部位中,12 个候选管家基因的Ct 值都处于15.09-28.70 之间,表明表达丰度比较适中。18S的Ct 值最小,介于15.09-16.15 之间,表明其基因表达丰度最高。GAPDH的Ct 值最大,处于24.55-28.70之间,表明其基因表达丰度最低。

图4 12 个管家基因的Ct 值分布箱线图Fig. 4 Ct value distribution box-plot of 12 house-keeping genes
2.5 候选管家基因在钩藤不同部位中表达稳定性分析
2.5.1 GeNorm 分析 将候选管家基因的原始Ct 值根据公式转化为相对表达量Q,再利用GeNorm 软件计算12 个候选管家基因表达稳定值M,通过M值来评估各个管家基因在钩藤不同部位的表达稳定性,M值越小表明候选管家基因的稳定性越高,反之越低。软件默认M值<1.5,表明基因相对稳定。GeNorm 软件分析结果显示(图5),在钩藤4 个部位中,12 个候选管家基因的稳定性排序为CYP=EF1-α>EF1-β>cdc73>18S>SAM>TUB>PAL>Acti n6>GAPDH>RNA L13>TUA。除RNA L13和TUA外,其余候选管家基因的M值都小于1.5,说明RNA L13和TUA表达稳定性差,不适合作为内参基因。而在不同部位中CYP和EF1-α 的M值最小,其表达最稳定。
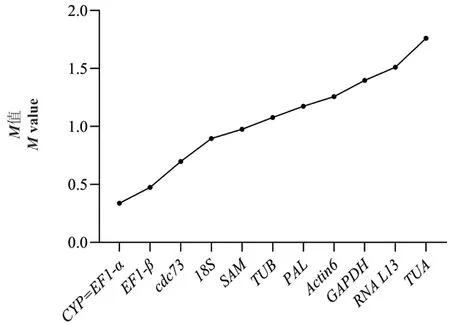
图5 12 个候选内参基因的GeNorm 分析Fig. 5 GeNorm analysis of 12 candidate reference genes
GeNorm 软件还可以通过计算变异值(Vn/n+1)来确定合适的内参基因数目。当Vn/n+1 <0.15 时,n个管家基因达到最适基因数目,反之则需要n+1 个。如图6 所示,V3/4的比值小于0.15,表明对于钩藤不同部位的最适管家基因数目为3 个。

图6 GeNorm 管家基因标准化因子配对变异值分析(Vn/n+1)Fig. 6 Standardization factor paired variation(Vn/n+1)analysis of house-keeping genes according to GeNorm
2.5.2 NormFinder 分析 与GeNorm 软件分析方法类似,将候选管家基因的原始Ct 值根据公式转化为相对表达量Q,再利用该软件计算12 个候选管家基因表达稳定值M,表达稳定值M与管家基因的表达稳定性呈负相关,即表达稳定值M越小,管家基因的表达稳定性越高,越适合作为管家基因。分析结果表明(图7),12 个候选内参基因表达稳定性依 次 为:SAM>18S>cdc73>EF1-β>PAL>TUB>CYP>Actin6>EF1-α>GAPDH> RNA L13>TUA, 即SAM的M值最小,为0.385,稳定性最高;
TUA的M值最大,为2.848,稳定性最差。

图7 12 个候选管家基因的NormFinder 稳定性分析Fig. 7 NormFinder stability analysis of 12 candidate housekeeping genes
2.5.3 BestKeeper 分 析 BestKeeper 软 件 不 同 于GeNorm 和NormFinder 软件,可直接利用RT-qPCR获得的Ct 值进行分析,计算Ct 值得到的标准偏差(SD)和变异系数(CV),从而判断候选管家基因的表达稳定性。该软件以SD=1 为阈值,若SD>1,则表明该基因表达不稳定,不适合作为管家基因。反之,SD 越小,说明基因表达越稳定;
CV 则反映出管家基因在不同部位中的表达水平变异程度,CV 越小,变异越小,越稳定。由表3 可知,SD 从小到大 依 次 为18S
表3 BestKeeper 分析12 个候选内参基因表达的稳定性Table 3 Expression stabilities of 12 candidate reference genes analyzed by BestKeeper
2.5.4 △Ct 分析 通过计算12 个候选管家基因的Ct 的平均标准偏差,来评估基因的表达稳定性。平均标准偏差越小,基因表达越稳定。△Ct 分析结果表明(图8),在钩藤4 个部位中,12 个候选管家基因的稳定性依次为SAM>18S>EF1-β>cd-c73>CYP>PAL>TUB>EF1-α> Actin6> GAPDH> RNA L13>TUA。即SAM基因的平均标准偏差最小,该基因表达最稳定。而TUA基因表达稳定性最差。

图8 12 个候选管家基因的△Ct 稳定性分析Fig. 8 △Ct stability analysis of 12 candidate house-keeping genes
2.5.5 管家基因稳定性综合分析 RefFinder 分析是根据每个候选管家基因在各程序中的结果分配适当的权重,然后通过计算所有算法的稳定值权重的几何平均值进行整体排序的稳定性综合评价的方法。RefFinder 分析方法避免了单个算法的片面性,能全面地分析候选管家基因的表达稳定性。通过RefFinder 计算出的稳定值,来评估基因的表达稳定性,稳定值越小,管家基因的稳定性越好。RefFinder 分析结果显示,在钩藤不同部位中管家基因的稳定性依次为SAM(3.08)>18S(3.16)>cdc73(4.76)>EF1-β(5.02)>CYP(6.00)>TUB(6.22)>PAL(6.86)>EF1-α(7.12)>TUB(7.44)>Actin6(7.96)>RNA L13(8.68)>GAPDH(9.12)>TUA(12.0)。因此,结合以上所有分析方法,在钩藤不同部位中,SAM的稳定值最高,其稳定性具有普适性,适合作为钩藤不同部位的管家基因。
2.6 钩藤生物碱上游途径相关基因表达分析
通过“转录组+表达谱+含量”共表达关联分析方法筛选大量候选基因,这种模式对于多基因或超基因家族候选序列的筛选具有很高的预见性,能够大大缩小后续基因研究范围。本课题组前期利用转录组数据发现了罗汉果甜苷V 生物合成通路中22 类基因,结合“转录组+表达谱+含量”共表达分析方法从80 条CYP450 和90 条UPDG 中筛选了可能参与罗汉果甜苷V 合成的7 个CYP450和5 个UDPG 候选酶基因[29],其中各有1-2 个基因通过原核表达及酵母得到了验证[30-31],共表达模式筛选重点候选基因对于提高其他物种的超基因家族目的基因的筛选效率具有重大的参考价值。以STR 基因为例(图9),总共从基因组中鉴定了22 条STR 候选基因,通过转录组、含量共表达分析,发现有3 条序列与钩藤碱和异钩藤碱含量聚为一类,从中挑选了g27792 基因作为重点序列进行了RT-qPCR 分析。本研究采用“基因组+转录组+代谢组”共表达关联分析的方式,同时对途径中其他基因进行筛选,这有效地缩小了基因功能验证的范围,提高了筛选效率。以SAM为管家基因,对钩藤不同部位中的钩藤生物碱上游途径的关键酶基因进行了表达模式分析。结果(图10)依次为环烯醚萜苷类途径中的Geraniol 8-hydroxylase(G8H,g4369)、8-hydroxygeraniol dehydrogenase(8-HGO,g27625)、Irid-oid synthase(IS, g8955)、Nepetalactol monooxygenase(7-DLS/CYP76A2, g24241)、7-deoxyloganetic acid glucosyltransferase(7-DLGT/UGT78A5,g32889)、7-deoxyloganate 7-hydroxylase(7-DLH/CY-P2A224,g31614)、Loganate methyltransferase(LAMT,g36536)、Secologanin synthase(SLS/CYP72A1,g43641);
色胺途径中的Anthranilate synthase(AS,g8946)、Anthranilate phosphoribosyltransferase(An-PRT, g21597)、Indole-3-glycerol phosphate synthase(IGPS, g14095)、Tryptophan synthase alpha chain(TSA,g3418)、Tryptophan synthase beta chain(TSB,g36741)、L-tryptophan decarboxylase(TDC,g28930)以及Strictosidine synthase(STR)在钩藤4 个部位的表达量。

图9 异胡豆苷合酶基因筛选热图Fig. 9 Screening heatmap of strictosidine synthase gene

图10 以SAM 为管家基因时钩藤生物碱生物合成上游途径中关键酶基因表达模式分析Fig. 10 Expression analysis of upstream pathway genes involved in TIAs biosynthesis using SAM as house-keeping gene
G8H、8-HGO、IS、7-DLS、7-DLGT、7-DLH基因的表达模式基本一致,在茎钩和叶片的表达量高,在根中的表达量最低。LAMT和SLS基因在叶片中的表达量明显高于其他部位。实验结果表明,AS、AnPRT、IGPS、TSA、TSB基因主要在蒴果中进行表达,其次是在茎钩和叶片中表达,在根中表达最少;
TDC基因主要在叶片中表达,在根中有微量表达,不在茎钩和蒴果中表达;
STR基因主要在蒴果中表达,其次在茎钩中表达,在根中表达量最少,这与钩藤碱和异钩藤碱在钩藤4 个部位中的含量变化呈现明显相关性,说明STR基因作为钩藤生物碱合成途径中的关键酶基因,调控着钩藤生物碱的含量。上游途径的量直接或间接决定终产物的产量,酶反应速率又决定了代谢途径的流量。
RT-qPCR 是分析基因表达模式的重要技术,也是药用植物次生代谢产物合成调控研究中必不可少的实验技术,而稳定的管家基因是获得准确实验结果的重要前提。植物的管家基因大部分具有维持细胞生命活动的功能,比如TUA、TUB、β2-MG、β-Actin是构成细胞器骨架的重要成分,GADPH、SAM、G6DPH、EF-1β则是参与植物体基本生化代谢过程。同一个管家基因在不同的实验条件、不同物种以及不同组织部位中稳定性具有差异。因此,要根据实际情况筛选最佳的管家基因。如SAM基因是花叶唐竹4 种叶色中表达最稳定的内参基因[32];
Actin和SAM则是研究赤霞珠葡萄发育后期基因表达的理想看家基因[33];
18S是在分析紫鸭跖草根系在不同铜浓度胁迫时表达最稳定的管家基因[34]。
本研究选取了18S、SAM、CYP、EF1-β、cdc73、TUB、EF1-α、PAL、Actin6、RNA L13、GAPDH、TUA这12 个植物中常见的管家基因。通过GeNorm、NormFinder、BestKeeper、ΔCT 以及RefFinder 综合评估候选管家基因分别在钩藤根、茎钩、叶片、蒴果中的稳定性。根据管家基因筛选结果显示,5 个软件所得结果基本一致,但也存在一定的差异,这可能是软件算法不同造成的。在钩藤的不同部位中,GeNorm 分析得出,CYP和EF1-α的稳定性最好;
而NormFinder 和 ΔCT 分析中SAM均是表达最稳定的管家基因;
而在BestKeeper 分析中,18S的稳定性最好,但SAM的SD、CV 值与18S差异不大,排名仅次于18S;
通过RefFinder 综合评估所有分析结果并进行稳定性排名,SAM是钩藤不同部位中最适的管家基因。综合以上5 款软件,SAM更适合作为钩藤的管家基因。
钩藤生物碱生物合成的上游途径分为环烯醚萜苷类途径和色胺途径。在环烯醚萜苷类途径中,在植物韧皮部表皮细胞中,G8H、8-HGO、IS、7-DLS、7-DLGT以及7-DLH参与了氧化、还原、糖基化及甲基化等反应,形成马钱子酸[35]。本研究发现,这些基因主要在茎钩和叶片中表达,可能是因为茎钩和叶片中韧皮部组织发达有关。随之马钱子酸在某种尚未阐明的机制下被运送到叶面表皮细胞[36],在LAMT 和SLS 的作用下生成断马钱子苷(secologanin),这与本研究的实验结果LAMT和SLS基因在叶片中的表达量最高一致。在色胺途径中,AS、AnPRT、IGPS、TSA、TSB基因主要在蒴果中进行表达,其次是在茎钩和叶片中表达,在根中表达最少。Noé 等[37]完成了 TDC 的首次分离纯化,该酶存在于植物细胞的细胞质中,只在叶片的上表皮细胞中表达,这与本实验结果一致,TDC基因在叶片上大量表达,在茎钩和蒴果中不表达。而STR基因主要在茎钩中表达,其次是蒴果,根中的表达量最低。这与钩藤碱和异钩藤碱在蒴果和茎钩中的含量高,叶片和根中含量低的趋势一致,证明该基因很可能是参与钩藤生物碱生物合成的STR候选基因。
本研究选取了18S、SAM、CYP、EF1-β、cdc73、TUB、EF1-α、PAL、Actin6、RNA L13、GAPDH、TUA这12 个植物中常见的管家基因,通过GeNorm、NormFinder、BestKeeper、ΔCT 以及RefFinder 综 合评估候选管家基因,最终筛选出SAM可作为钩藤不同部位RT-qPCR 分析时最合适的管家基因。对钩藤生物碱生物合成上游途径中关键酶基因进行筛选,以SAM为看家基因对这些基因在钩藤不同部位的表达模式进行分析,结果表明这些关键酶基因在钩藤不同部位中的表达具有特异性,其变化趋势与含量趋势基本一致,可能参与钩藤生物碱生物合成途径。
猜你喜欢钩藤生物碱引物DNA引物合成起始的分子基础广东药科大学学报(2022年3期)2023-01-04高中生物学PCR技术中“引物”相关问题归类分析生物学通报(2022年1期)2022-11-22不同产地钩藤的药用成分含量及不同温度烘干的影响农技服务(2021年7期)2021-09-24HPLC法同时测定痹通药酒中4种生物碱成分中成药(2017年12期)2018-01-19HPLC-Q-TOF/MS法鉴定血水草中的异喹啉类生物碱中成药(2017年7期)2017-11-22HPLC-Q-TOF/MS法鉴定两面针和单面针中的生物碱中成药(2017年8期)2017-11-22呼吸道感染样本中IFITMs引物优化罕少疾病杂志(2017年2期)2017-02-23火炬松SSR-PCR反应体系的建立及引物筛选广西林业科学(2016年3期)2016-03-16对叶百部中非生物碱化学成分的研究中国当代医药(2015年24期)2015-03-01HPLC测定钩藤药材中钩藤碱、异钩藤碱的含量中国民族民间医药(2012年18期)2012-11-20